通过将语言模型应用于蛋白质与药物的相互作用,研究人员可以快速筛选大量的潜在药物化合物库。巨大的药物化合物库可能拥有治疗各种疾病的潜力,如癌症或心脏病。理想情况下,科学家们希望通过实验对这些化合物中的每一种针对所有可能的目标进行测试,但进行这样的筛选是非常耗时的。
keyimage.jpg© 由 cnBeta.COM 提供
近年来,研究人员已经开始使用计算方法来筛选这些化合物库,希望能加快药物发现的速度。然而,其中许多方法也需要很长的时间,因为它们中的大多数都是从氨基酸序列中计算出每个目标蛋白的三维结构,然后用这些结构来预测它将与哪些药物分子相互作用。
麻省理工学院和塔夫茨大学的研究人员现在已经设计出一种基于一种被称为大型语言模型的人工智能算法的替代计算方法。这些模型--一个著名的例子是ChatGPT--可以分析大量的文本,并找出哪些词(或者,在这种情况下是氨基酸)最有可能一起出现。这个被称为ConPLex的新模型可以将目标蛋白质与潜在的药物分子相匹配,而不必执行计算分子结构的密集步骤。
使用这种方法,研究人员可以在一天内筛选出超过1亿个化合物--比任何现有模型都要多。
麻省理工学院计算机科学与人工智能实验室(CSAIL)计算与生物学组组长、西蒙斯数学教授邦尼-伯杰(Bonnie Berger)说:"这项工作解决了对潜在候选药物进行高效和准确的硅计算筛选的需求,而且该模型的可扩展性使得大规模筛选可以评估脱靶效应、药物再利用以及确定突变对药物结合的影响。"
塔夫茨大学计算机科学教授Lenore Cowen也是该论文的资深作者,该论文于6月8日发表在《美国国家科学院院刊》上。CSAIL的研究科学家Rohit Singh和麻省理工学院的研究生Samuel Sledzieski是这篇论文的主要作者,麻省理工学院生物工程副教授、麻省理工学院和哈佛大学Ragon研究所成员Bryan Bryson也是作者。除了这篇论文,研究人员还在网上提供了他们的模型供其他科学家使用。
pnas.2220778120fig02.jpg© 由 cnBeta.COM 提供
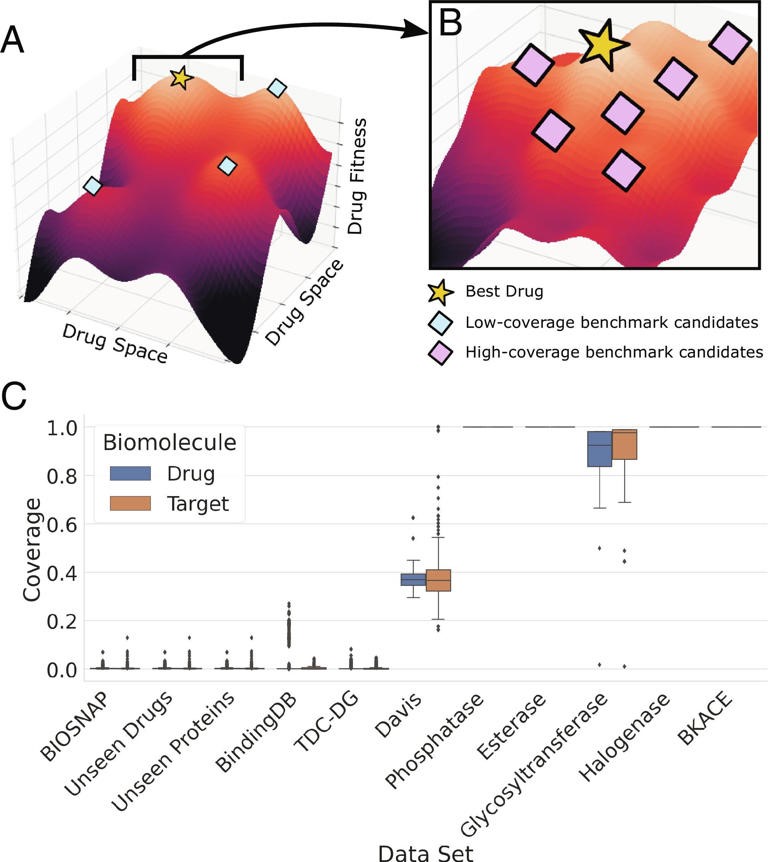
近年来,计算科学家在开发能够根据蛋白质的氨基酸序列预测其结构的模型方面取得了巨大的进展。然而,使用这些模型来预测大型潜在药物库如何与一个癌症蛋白质相互作用,例如,已被证明具有挑战性,主要是因为计算蛋白质的三维结构需要大量的时间和计算能力。
另一个障碍是,这类模型在消除被称为诱饵的化合物方面没有良好的记录,这些诱饵与成功的药物非常相似,但实际上并不能与目标发生良好的互动。
Singh说:"该领域的一个长期挑战是这些方法是脆弱的,也就是说,如果我给模型一种药物或一种小分子,看起来几乎像真正的东西,但它在某些微妙的方面略有不同,该模型可能仍然预测它们会相互作用,尽管它不应该。"
研究人员已经设计出了能够克服这种脆弱性的模型,但它们通常只针对一类药物分子,而且由于计算时间过长,它们并不适合大规模筛选。
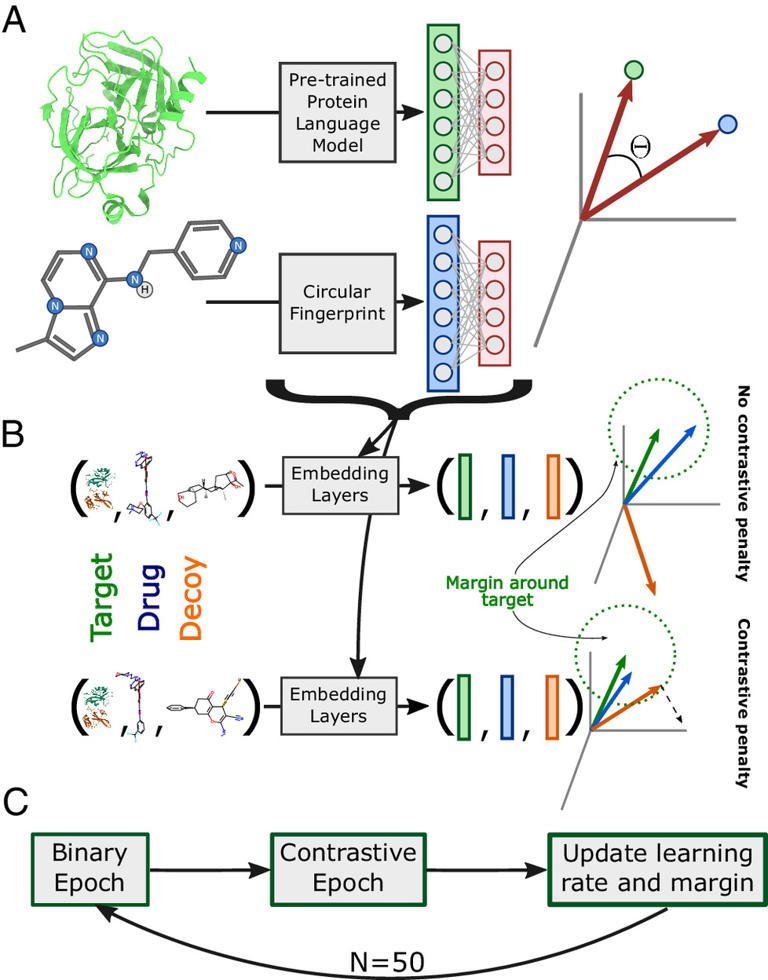
麻省理工学院的团队决定采取另一种方法,基于他们在2019年首次开发的一个蛋白质模型。与一个包含2万多个蛋白质的数据库合作,该语言模型将这些信息编码为每个氨基酸序列的有意义的数字表示,这些数字表示捕捉了序列和结构之间的关联。
Sledzieski说:"有了这些语言模型,即使是序列非常不同但可能具有类似结构或类似功能的蛋白质也可以在这个语言空间中以类似的方式表示,我们能够利用这一点来进行预测。"
在他们的新研究中,研究人员将蛋白质模型应用于找出哪些蛋白质序列将与特定药物分子相互作用的任务,两者都有数字表示,通过神经网络转化为一个共同的共享空间。他们对已知的蛋白质-药物相互作用进行了训练,这使得它能够学会将蛋白质的具体特征与药物结合能力联系起来,而不必计算任何分子的三维结构。
"有了这种高质量的数字表示,该模型可以完全绕过原子表示,并从这些数字中预测这种药物是否会结合,"Singh说。"这样做的好处是,你避免了通过原子表示法的需要,但这些数字仍然有你需要的所有信息。"
这种方法的另一个优点是,它考虑到了蛋白质结构的灵活性,当与药物分子相互作用时,蛋白质结构可能是"摇摆不定"的,并呈现出略微不同的形状。
高亲和力
为了使他们的模型不太可能被诱饵药物分子所愚弄,研究人员还纳入了一个基于对比学习概念的训练阶段。在这种方法下,研究人员给模型提供了"真实"药物和诱饵的例子,并教它区分它们。
然后,研究人员通过筛选大约4700个候选药物分子库来测试他们的模型,看它们是否能与一组被称为蛋白激酶的51种酶结合。
研究人员从排名靠前的药物中选择了19个药物-蛋白对进行实验测试。实验显示,在这19个命题中,有12个具有很强的结合亲和力(在纳摩尔范围内),而几乎所有其他可能的药物-蛋白质配对都没有亲和力。这些配对中的四个以极高的、亚纳摩尔的亲和力结合(如此之强,以至于极小的药物浓度,即十亿分之一,就能抑制该蛋白质)。
虽然研究人员在这项研究中主要侧重于筛选小分子药物,但他们现在正致力于将这种方法应用于其他类型的药物,如治疗性抗体。这种建模也可以证明对潜在的药物化合物进行毒性筛选是有用的,以确保它们在动物模型中测试之前没有任何不必要的副作用。
"药物发现如此昂贵的部分原因是它有很高的失败率。"Singh说:"如果我们能够通过预先说这种药物不可能成功来减少这些失败率,这可以在很大程度上降低药物发现的成本。"
美国国家癌症研究所癌症数据科学实验室主任Eytan Ruppin说,这种新方法"代表了药物-靶点相互作用预测的重大突破,并为未来的研究提供了更多机会,以进一步提高其能力",他并没有参与这项研究。"例如,将结构信息纳入潜伏空间或探索生成诱饵的分子生成方法可以进一步改善预测。"
免责声明:
文章系本网编辑转载,会尽可能注明出处,但不排除无法注明来源的情况,转载目的在于传递更多信息,并不代表本网赞同其观点和对其真实性负责。如涉及作品内容、版权和其它问题,请在30日内与本网联系.
[声明]本站文章版权归原作者所有,内容为作者个人观点,不代表本网站的观点和对其真实性负责,本站拥有对此声明的最终解释权.